Vivant
- Santé, Médecine et Sciences du Vivant
- Biologie & Biochimie
Des protéines anti-grippe créées par ordinateur
- Tweeter
-
-
0 avis :
Le vaccin universel contre le virus de la grippe n'existe pas, notamment parce que chaque année, les souches du virus évoluent et changent. L'objectif des scientifiques est de trouver une molécule unique qui pourrait stopper n'importe quel virus. Cet exploit semble encore hors de portée avec les méthodes traditionnelles de criblage de nombreuses protéines pour trouver celle qui neutraliserait le plus de souches virales. Sarel Fleishman, de l'Université de Washington à Seattle, et ses collègues ont donc développé une méthode informatique pour identifier la molécule efficace contre tous les virus de grippe ; à partir de leurs résultats, ils ont ensuite créé deux protéines susceptibles de neutraliser n'importe quel virus de la grippe. Reste maintenant à les produire en grande quantité.
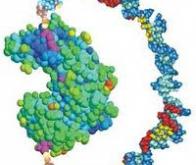
La surface des virus de la grippe comporte des protéines nommées hémagglutinines qui permettent aux virus d'infester les cellules et notamment les voies respiratoires. Ces hémagglutinines sont souvent les cibles des thérapies antivirales et des anticorps (les molécules de défense de l'organisme), car elles sont essentielles à la virulence. Elles possèdent deux régions : la région externe, très variable d'une souche de virus à l'autre, est la cible des anticorps et des vaccins ; la région basale est quant à elle très conservée entre les différentes souches, mais elle est peu accessible. Il est donc difficile d'identifier une protéine ou un anticorps qui pourrait se fixer sur cette région et ainsi neutraliser le virus.
Les biophysiciens sont donc partis de ce domaine conservé des hémagglutinines, dont on connaît l'organisation des atomes et la structure tridimensionnelle, et ont appliqué un protocole original. Ils ont d'abord identifié les « points chauds » du domaine, c'est-à-dire les régions qui interagissent avec des acides aminés isolés : ils ont testé un par un divers acides aminés et conservé ceux qui se fixaient le plus favorablement (avec la meilleure énergie de liaison) sur le domaine des hémagglutinines. Puis ils ont cherché dans une banque de protéines celles possédant les acides aminés des points chauds et dont le repliement tridimensionnel serait le plus complémentaire de l'hémagglutinine (à la manière d'une clé dans une serrure).
En utilisant des programmes développés par une vingtaine d'équipes de recherche dans le monde et plus de 100 000 heures de calculs, les chercheurs ont résolu ce puzzle complexe et créé par ordinateur des protéines complètes capables d'interagir fortement et spécifiquement avec le domaine conservé des hémagglutinines.
Ensuite, par génie génétique, S. Fleishman et ses collègues ont fait produire ces protéines à la surface de levures (en introduisant dans leur génome les séquences d'ADN correspondant aux cyberprotéines obtenues par ces chercheurs). Ils en ont trouvé deux qui présentent une affinité de l'ordre du nanomolaire avec le domaine conservé des hémagglutinines : en d'autres termes, si l'on place un milliard de ces protéines en solution avec des hémagglutinines, seule une reste libre, les autres étant liées à une hémagglutinine. Ces deux protéines sont très proches de celles conçues par ordinateur, à quelques mutations près.
Enfin, les chercheurs ont confirmé leurs résultats en obtenant par analyse aux rayons X la structure tridimensionnelle de l'une de ces protéines : la surface de contact observée entre l'hémagglutinine et la protéine conçue par ordinateur est très proche de celle prévue par la théorie. Ainsi, grâce à une élégante conception par ordinateur et sans recourir à des homologies avec des protéines déjà connues, les scientifiques ont découvert deux protéines capables de se lier avec une forte affinité à la région conservée des hémagglutinines. Les résultats obtenus par d'autres équipes et d'autres programmes antérieurs n'aboutissaient pas à des molécules présentant d'aussi fortes interactions. La prochaine étape consistera à produire ces protéines en grande quantité et à déterminer leur efficacité pour lutter contre le virus.
Noter cet article :
Vous serez certainement intéressé par ces articles :

Maladie de Charcot : un nouveau traitement prometteur dévoilé aux États-Unis
Des chercheurs américains ont annoncé un nouveau traitement plein de promesses contre la maladie de Charcot, mortelle et pour l’instant sans cure efficace connue. Celle qui est aussi appelée ...

Une technique inspirée des papillons pour détecter le cancer
Des scientifiques de l'Université de l'Illinois Urbana-Champaign ont développé une technologie de détection du cancer en s'inspirant du système visuel avancé des papillons. Cette nouvelle ...
Vers un test ARN plus précis et plus fiable pour détecter le cancer du côlon
Le cancer colorectal (CCR) est le 3e cancer le plus fréquent chez l’homme et le 2e chez la femme, et entraîne plus de 17 000 décès/an en France. La survie à 5 ans de ce cancer est bien plus élevée ...
Recommander cet article :
- Nombre de consultations : 50
- Publié dans : Biologie & Biochimie
- Partager :