Vivant
- Santé, Médecine et Sciences du Vivant
- Biologie & Biochimie
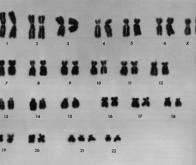
L'organisation tridimensionnelle des chromosomes enfin cartographiée
- Tweeter
-
-
0 avis :
Les chercheurs s'en doutaient depuis quelques années déjà : un individu est plus que la somme de ses gènes ; l'architecture de ses chromosomes serait aussi un facteur clé pour le bon fonctionnement de ses cellules. Mais jusqu'ici, une réelle compréhension des principes qui organisent le repliement du génome dans le noyau échappait aux analyses. Et depuis 4 ans, une vingtaine d'équipes dans le monde tentaient de mettre au point une technique d'analyse pour étudier ce repliement.
La méthode de l'équipe CNRS dérive d'une autre développée par une équipe américaine en 2009. Une technique baptisée « capteur de la conformation des chromosomes» (Hi-C), qui permet de décrire les contacts que chaque région du génome établit avec les autres. Comme la version américaine, la méthode montpelliéraine consiste en trois grands étapes : figer les contacts dans ou entre les chromosomes avec le composé formaldéhyde ; lier les bouts des deux gènes en contact avec la molécule ADN ligase, d'origine bactérienne ; et déterminer la séquence de ces deux gènes. Mais Giacomo Cavalli et ses collègues y ont apporté d'importantes améliorations rendant possible l'étude de fragments d'ADN beaucoup plus petits (de quelques centaines de paires de bases, contre des fragments de 5-10 kilobases avant). Ce qui permet de cartographier ces contacts à haute résolution.
Avec cette technique, les chercheurs montpelliérains et leurs collègues israéliens ont analysé le génome de la mouche du vinaigre ou drosophile. Un animal avec un génome quinze fois plus petit que le nôtre. Résultat : l'équipe a pu y identifier… 118 millions de zones de contact ! L'analyse de ces zones leur a permis ensuite d'établir des cartes d'interaction très détaillées. Ces cartes montrent que les chromosomes sont organisés en domaines contenant un ou plusieurs gènes. Séparés par des régions frontières, ces domaines correspondent soit à des zones activement exprimées, soit à des régions inactives.
Grâce aux compétences en mathématiques de l'équipe israélienne de Amos Tanay, les deux équipes ont pu ensuite développer un modèle informatique capable de prédire assez fidèlement le repliement réel des chromosomes. Très puissant, cet outil a permis de décrypter les principes fondamentaux qui régissent l'organisation tridimensionnelle du génome. Notamment, il est apparu que les domaines inactifs ont tendance à former des contacts avec d'autres domaines inactifs souvent proches sur le même chromosome, alors que les domaines actifs « contactent » d'autres domaines actifs lointains ou proches et éventuellement sur d'autres chromosomes.
Cruciaux, ces travaux devraient permettre de mieux comprendre les différences de repliement chromosomiques selon les types de cellules (cellules de foie, de muscle, nerveuse, etc.) et l'importance de ce repliement dans la survenue de cancers.
Noter cet article :
Vous serez certainement intéressé par ces articles :
Découverte de deux molécules-clé régulant le processus de vieillissement
Deux équipes de recherche indépendantes, l'une canadienne et l'autre américaine, viennent de découvrir deux nouveaux mécanismes biologiques fondamentaux qui joueraient un rôle-clé dans le processus ...

Des omégas 3 pour prévenir les risques associés à l'obésité
Selon l’OMS, depuis 1975, l’obésité a presque triplé à l’échelle planétaire et atteint 650 millions de personnes. La pathologie est associée à de nombreuses comorbidités (diabète de type 2, maladies ...

Un premier traitement contre le vitiligo disponible en France
Ce premier traitement contre le vilitigo, dont le CHU de Bordeaux a participé à l’essai clinique, remboursé par la CPAM, répond enfin au besoin des patients qui souffrent de cette maladie très ...
Recommander cet article :
- Nombre de consultations : 250
- Publié dans : Biologie & Biochimie
- Partager :